Evolution: Mehr Komplexität, weniger Tyrosin
BLOG: Fischblog

Als vor ein paar Jahren die ersten vollständigen Genome entschlüsselt waren, stellten Wissenschaftler zu ihrer Verwunderung fest, dass die Größe eines Genoms wenig mit der Komplexität des zugehörigen Organismus zu tun hat. Manche Einzeller haben zigfach mehr Erbgut als selbst die komplexesten Säugetiere. Gibt es überhaupt biochemische Merkmale, die eindeutig mit Komplexität zusammen hängen und möglicherweise Aussagen über ihre Ursachen zulassen? Wissenschaftler sind dieser Frage nachgegangen und auf eine sehr merkwürdig erscheinende Antwort gestoßen: Offenbar hängt die Komplexität von Organismen systematisch mit dem Anteil der Aminosäure Tyrosin in den Proteinen zusammen.
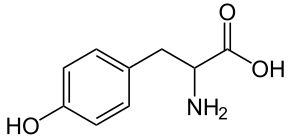
Das ergibt zuerst einmal keinen Sinn, denn Tyrosin[1] ist eigentlich eine Allerwelts-Verbindung. Sie gehört weder zu den häufigsten noch den seltensten Aminosäuren in Organismen und kann vom Körper problemlos selbst produziert werden. Immerhin ist sie unter anderem Ausgangsstoff für die Biosynthese des Haut- und Haarpigments Melanin und des Adrenalins. In den Proteinen allerdings spielt sie keine Rolle, die sie gegenüber den ähnlichen Aminosäuren wie Phenylalanin auszeichnen würde. Trotzdem gibt es einen klaren Zusammenhang: Je komplexer der Bauplan einer Art, desto weniger Tyrosin enthalten ihre Proteine.
Das kann verschiedene Ursachen haben. Die Biosynthesekosten für Tyrosin sind vergleichsweise hoch, so dass die Zelle hier Energie einsparen könnte. In dem Fall müsste sich ein ähnlicher Effekt allerdings auch bei den noch teureren Aminosäuren Tryptophan und Phenylalanin nachweisen lassen. Tatsächlich nimmt der Tryptophan-Anteil mit zunehmender Zahl unterschiedlicher Zelltypen tendenziell zu.
Selektionseffekt durch Regulationsenzyme
Die Autoren der Veröffentlichung schlagen daher eine andere Erklärung vor. Sie vermuten, dass es sich um einen Selektionseffekt handelt, der von Tyrosin-Kinasen verursacht wird. Diese Biomoleküle knüpfen einen Phosphatrest an die Hydroxy-Gruppe des Tyrosins. Diese chemische Veränderung beeinflusst die Aktivität des betreffenden Enzyms. Auf diese Weise können Tyrosinkinasen ganze Stoffwechselwege ein- und ausschalten, und so sind sie an vielen grundlegenden zellulären Prozessen beteiligt. Neben der Zellteilung (was sie interessant für die Krebsforschung macht) sind sie besonders wichtig für die Differenzierung, also der Entwicklung einer unreifen Stammzelle zu einem ganz bestimmten Zelltyp.
Die Tyrosin-Kinasen haben nun eine ganz naheliegende Verbindung zur Komplexität: Je mehr unterschiedliche Zelltypen ein Organismus hat, desto mehr Signalwege braucht er, um ihre Entwicklung einzuleiten. Und das bedeutet auch: Mehr Tyrosinkinasen. Tatsächlich findet man einen solchen Zusammenhang.
Dass gleichzeitig mit zunehmender Anzahl der Tyrosin-Kinasen immer weniger Tyrosin in Proteine eingebaut wird, erklären die Forscher folgendermaßen: Irgendwann auf dem Weg zur Vielzelligkeit entstand die erste Tyrosin-Kinase, aus der alle anderen Enzyme dieser Klasse durch Genverdoppelung und Mutationen hervorgegangen sind. Jede auf diese Weise neu entstandene Kinase phosphoryliert nun nicht nur ein einziges Tyrosin an einer ganz bestimmte Stelle, sondern – mit deutlich geringerer Effizienz – auch andere, an die sie nicht heran soll.
Es gibt also neben der erwünschten, also durch die Evolution positiv selektierten Reaktion einer Kinase ein “Hintergrundrauschen” an ähnlichen Reaktionen, die nur Energie kosten oder gar Schaden anrichten. Und es gibt zwei Wege, das Hintergrundrauschen zu minimieren. Der eine ist, die Kinase noch spezifischer auf die eine erwünschte Reaktion zuzuschneiden. Das kann, da die Evolution ein zufallsgesteuerter Prozess ist, ziemlich lange dauern. Wenn überhaupt.
Man kann die unerwünschten Reaktionen aber auch einfach unterdrücken, indem man einfach alle potentiellen falschen Reaktionspartner aus dem Verkehr zieht. Und genau das scheint hier im Laufe der Zeit zu geschehen. Tatsächlich ist der Effekt nicht nur auf Organismen-Ebene zu sehen, sondern sogar bei den Proteinen selbst. Biomoleküle, die von Tyrosin-Kinasen phosphoryliert werden, enthalten wesentlich weniger Tyrosin als solche Enzyme, die kein Ziel dieser Reaktion darstellen – offensichtlich ist das Risiko besonders hoch , dass Tyrosin-Kinasen im richtigen Zielmolekül das falsche Tyrosin erwischen.
Komplexität durch posttranslationale Modifikation
Die Zelle entfernt also im Laufe der Zeit Tyrosin aus den Proteinen. Das darf aber nicht völlig zufällig passieren, denn oft sind die besonderen Eigenschaften der Aminosäure wichtig für die Struktur des Proteins. Tauscht man zum Beispiel eine Saure Aminosäure gegen eine basische, verändert sich die Ladung an diesem Ort und das Protein wird sich möglicherweise ganz anders falten. Tyrosin ist eine aromatische Aminosäure und sollte deswegen durch eine andere aromatische Aminosäure, also entweder Phenylalanin oder Tryptophan, ersetzt werden. Dementsprechend sollte deren Anteil im Genom parallel zur Anzahl Tyrosin-Kinasen ansteigen. Tatsächlich ist das für Tryptophan der Fall.[2]
Interessant ist, dass man einen ähnlichen Effekt auch bei anderen Aminosäuren beobachten kann, die nachträglich durch Enzyme verändert werden.[3] Diese Ergebnisse deuten darauf hin, dass es gerade die nachträgliche Veränderung von Proteinen ist, die den enormen Anstieg der Komplexität bei vielzelligen Organismen erst ermöglicht hat. Die genaue Erforschung solcher posttranslationaler Modifikationen, und hier speziell die Glycoproteine und Proteoglycane, wird Biologie und Medizin im 21. Jahrhundert in einem ähnlichen Maße revolutionieren wie es der genetische Code und das zentrale Dogma der Molekularbiologie es getan haben.
.
.
.
[1]Die Stereochemie im Bild bitte dazudenken.
[2] Eine mögliche Erklärung bietet der genetische Code: während die Codons fürTyrosin und Tryptophan in der zweiten Position beide eine Purinbase haben, müsste man für Phenylalanin die Purinbase Adenin gegen die Pyrimidin-Base Uracil austauschen (die dritte Position ist dabei weniger kritisch).
[3] Außer bei Serin, kurioserweise.
–
Tan, C., Pasculescu, A., Lim, W., Pawson, T., Bader, G., & Linding, R. (2009). Positive Selection of Tyrosine Loss in Metazoan Evolution Science, 325 (5948), 1686-1688 DOI: 10.1126/science.1174301


Anmerkung zu den Diagramm-Beschriftungen:
Zur Orientierung:
S.cer = Saccharomyces cerevisiae, Bierhefe
C.ele = Caenorhabditis elegans, Fadenwurm
D.mel = Drosophila melanogaster, Taufliege
X.tro = Xenopus tropicalis, Krallenfrosch
R.nor = Rattus norvegicus, Ratte
M.mus = Mus musculus, Hausmaus
C.fam = Canis familiaris, Hund
B.tau = Bos taurus, Hausrind
P.tro = Pan troglodytes, Schimpanse
H.sap = Homo sapiens, Mensch
Die anderen müsst ich nachgucken, aber ich denke die Richtung ist erkennbar.
Ich habe das Paper zwar noch nicht gelesen, aber möchte trotzdem etwas zu diesem Thema sagen: Unter der Komplexität der DNA versteht man doch die Gesamtlänge der DNA, die aus nicht repetitiven Sequenzen besteht. Sprich die Abschnitte bzw. Gene, die wirklich exprimiert werden. Ich Frage mich jetzt, was die AS Tyrosin mit der Komplexität zu tun haben soll? Ich glaube eher, dass die Wissenschaftler darauf hinaus wollten zu sagen, dass ein Organismus mehr Proteine herstellt als das Gene vorhanden sind. Dass das auf die posttranslationale Modifikationen von Proteinen zurückzuführne ist, ist ja ganz klar! Ein Hintergrundrauschen ist bei vielen Reaktionen zu finden und daher nix besonderes. Das mit den Tyrosin-Kinasen hab ich noch nicht ganz verstanden. Soll die Aussage sein, dass je komplexer ein Organismus ist, desto mehr verschiedene Zellen hat er und desto mehr Tyrosin-Kinasen braucht er, um diese Gewebe zu produzieren?
Von der Komplexität der DNA ist weder im Paper noch in diesem Text die Rede.
*Ich glaube eher, dass die Wissenschaftler darauf hinaus wollten zu sagen, dass ein Organismus mehr Proteine herstellt als das Gene vorhanden sind.*
Nein. Das ist nicht neu, dann hätte man sich das Paper auch sparen können.
*Soll die Aussage sein, dass je komplexer ein Organismus ist, desto mehr verschiedene Zellen hat er und desto mehr Tyrosin-Kinasen braucht er, um diese Gewebe zu produzieren?*
Das ist die Hypothese.
“Dass das auf die posttranslationale Modifikationen von Proteinen zurückzuführne ist, ist ja ganz klar! “
Ich bin schon eine Weile raus aus dem Fach, müsste es aber nicht korrekterweise die posttranskriptionelle Modifikation heissen (alternatives splicen etc.)?
Gruß